
出品 | 新浪科技《科学大家》
撰文 | Stefan Hell 德国物理学家、马克斯·普朗克生物物理化学研究所所长,2014年诺贝尔化学奖得主
整个20世纪,科学家始终认为光学显微镜的分辨率不可能超过200纳米。也就是说,只要两点之间的距离小于200纳米,用光学显微镜便无法分辨清楚。但随着21世纪的到来,有关研究揭示,这个分辨率极限其实是可以跨越并解决的。STED显微镜:受激发射损耗显微镜就完美地解决了这一问题。它利用共聚焦可见光获得荧光物质的高分辨率显微图像,大大地改变了光学显微镜的观察能力。后来随着有更高分辨率的光学显微镜被研发出来,现在的标准分辨率已经达到了20纳米,远高于之前的200纳米。因研制出超分辨率荧光显微镜,2014年,Eric Betzig、William E。 Moerner我们三个获得了诺贝尔化学奖。

STED显微镜运作原理
所有超分辨率显微镜的关键都是用染料给样本染色。比如先用一道绿色光束激活20个分子,这个叫做激发光束;再用另一道光束使部分分子停止发光,这个叫抑制光束。用这种经过特意选择的方式,便可以实现更高的分辨率。
上世纪90年代末,我和我的研究团队还拿不准这项研究能否成功,因为它看上去实在太复杂了。但了解了它的运作原理之后,就显得简单多了。等简化到一定程度后,就可以实现商业化。如今STED显微镜已经实现了商业化,可以安装在任何品牌的镜架上,使用非常便捷。目前,已经有人发表了对STED显微镜的使用评述。比如在生物分子领域,它可以用来观察线粒体等复杂细胞器的结构,可以看清线粒体中的结构分布,而此前人们是看不到这些的。
神经科学领域也是STED显微镜的一大重要应用领域。研究人员用STED显微镜观察了神经元的细胞骨架。哈佛大学庄小威教授还将该技术扩展运用到了活细胞成像领域,她发明的随机光学重建显微技术,这项技术的优势就在于可以实现活细胞成像,既实现了高分辨率,又能在活性环境中进行。在最极端的情况下,活细胞成像可以用于观察活生物体的活组织。
近期,我们发表了一篇论文,以40至50纳米的空间分辨率、对小鼠活体突触的蛋白质分布做了成像处理。这项成果很令人激动,因为这意味着,我们能够观察到生物活体中突触的蛋白质分布。除此之外,该技术还能做的更多。我们可以用高分辨率显微镜、尤其是STED显微镜来解决研究中遇到的问题。但我是一名物理学家,这些领域并不是我的专长。不过我也想尽力帮助大家,如果我能发明一套系统、找到某种解决方案、或者提出某个物理概念,能够对现状有所改变,那么当我的理论发展成熟后,就应该交给企业进行商业化,确保理论能投入实际应用。
我认为,如果你对活细胞、活神经元、甚至活生物体的亚衍射分辨率成像感兴趣的话,STED将对你非常有用。STED与其它高分辨率显微镜的区别在于,它的工作方式类似于共聚焦显微镜,可以和共聚焦显微镜无缝融合。你不需要担心染料浓度、后期数学处理等问题,可以把注意力集中在要研究的生物学问题上。所以我相信这项技术会变得非常流行。之前这台显微镜还比较贵,但现在已经稍微降了一点。随着科技进步,很多一开始非常复杂的东西到最后都会变得很简单。

2014年诺贝尔化学奖官方海报
不过我不是研究显微镜的应用的,刚才也提到,我是一名物理学家。不过我一直很希望能突破衍射极限、实现更高的分辨率。上图是2014年诺贝尔化学奖的官方海报,上面突出了STED和PALM/STORM显微镜。虽然两者之间存在区别,但也有很多共同之处。比如说,这两种显微镜在原则上都能达到与原子直径相当的分辨率,但在实际中却达不到。所以在获得诺贝尔奖时,这两种技术都尚未实现几纳米级的空间分辨率。不过有一些例外情况:在一些特殊条件下,对于一些特殊分子,也可以实现更高的空间分辨率。但整个记录过程一般要持续数小时,但仅适用于观察特定领域,还不能推广到更多的领域。
所以在获得诺奖后,我问了自己一个问题——就像诺贝尔奖委员会说的那样,这些技术成功跨越了分辨率门槛,但还没有达到最终极限。于是我将这个难题作为新的目标,去解决它。
虽然获得了诺奖,但只跨越了初级门槛,这是远远不够的,我们还要达到最终极限才行。所以接下来我要谈谈在获得诺奖之后,在分辨率方面取得的进展,我将其称作“后诺奖超分辨率”。
后诺奖超分辨率时代

这个概念叫MINFLUX,论文大约在一年前发表。该技术具有把分辨率提高至1纳米的潜力。我现在要讲讲如何把分辨率提高至1纳米。
首先解释一下超分辨率显微镜究竟是如何运作的。现在有各种各样的解释,但相信我,其中90%、甚至95%都是错的,或者有误导性。只有一种解释还算能自圆其说。它其实很简单,一旦你理解了之后,就会深受启发。
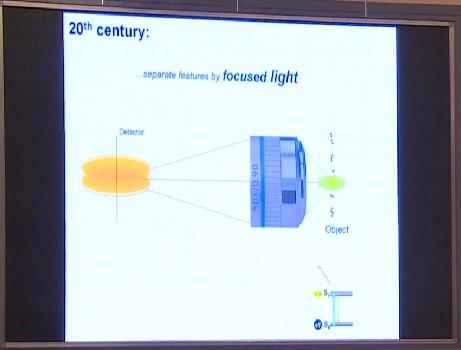
之前的显微镜为何无法突破衍射极限
为何20世纪的显微镜无法突破衍射极限、实现更高的分辨率呢?答案很简单,因为所有的显微镜,都试图用聚焦光把要观察的特征区给分开。这些显微镜都安装了透镜,均可以生成一束聚焦光。如果把这束光控制得很细,照亮的分子就很少,就能区分更细节的特征。但你们都在学校里学过,受光线衍射的影响,这种细节是有极限的。而如果照亮整个视野,只对其中发出荧光的分子做成像处理,依然会受到衍射的影响,会产生光晕,从而对分辨率造成限制。如果这一范围内包含很多特征,看上去就会重叠在一起、无从分辨。换句话说,你只要用光线聚焦来区分特征,就会遇到衍射极限的问题。
而我们超分辨率显微镜的根本思想就是,不用光线来区分特征,尤其是不用聚焦光,而是用分子态来区分。一旦你改变了要观察的分子状态,就不再受光线聚焦和光晕大小的制约了。这就是STED、PALM/STORM等显微镜的基本原理。我们改变分子态,就是为了把特定分子与周围的分子区分开来。简单来说,就像黑与白那样分明。分子态分为基态与激发态。绿色的激发光束可以把荧光分子从“关闭”状态切换成明亮的“发光”状态。这样一来就能把某个分子与相邻分子区分开,从而获得更高的分辨率。
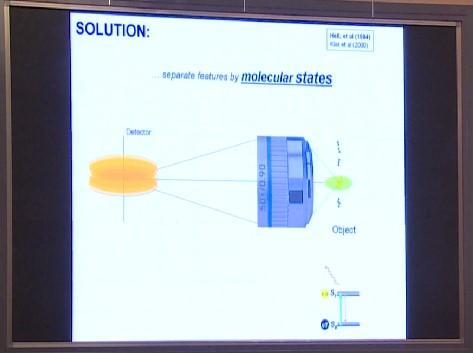
那我们如何对绿色光斑中的分子进行区分呢?答案很简单。只要把除中央分子之外的分子“关闭”、让其不再发光就行。要把周围的分子关闭,我们可以发射一束甜甜圈状的光线,中间留出一个洞,这样就能把除中央分子外的分子都“关掉”了,让其它分子从荧光态变回基态。这就是STED显微镜的基本原理,通过发射第二道光束,让其它分子都停止发光,只保留中间分子的荧光。经过一段时间的测量后,就可以由探测器将它们区分开来了。只要对所有分子进行逐个扫描,最终就能把所有特征区分清楚。
STED、PALM/STORM显微镜有何区别
运用这道光束,我们可以决定哪些分子处于“发光”状态,哪些处于“关闭状态”,从而可以分清不同分子的位置。探测器会探测分子发出的荧光,然后做成像处理。
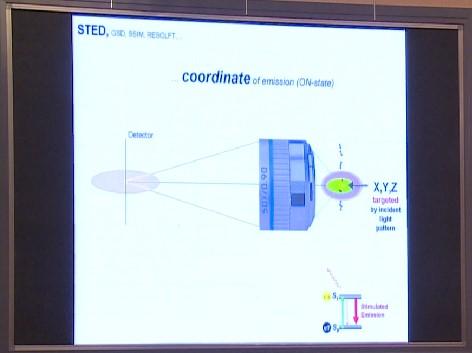
这个过程不仅可以用来启动受激发射,还可以实现不同分子态之间的切换。这些举措都可以实现更高的分辨率。但正如我刚才所说,关键还在于分子“关闭”和“发光”状态的区别,只要能决定哪些分子“关闭”、哪些分子“发光”,你就能确定相应分子的位置了。
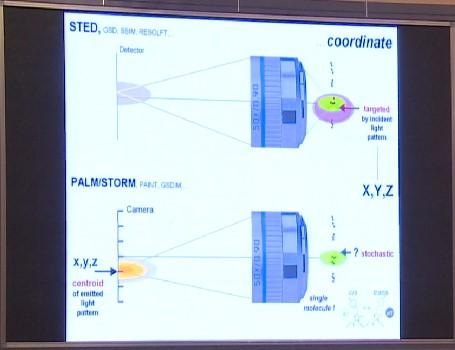
刚才说的都是STED显微镜,现在再来看看PALM/STORM显微镜,它们的发明时间比STED晚了六年。与STED显微镜相比,PALM/STORM显微镜有什么区别呢?区分特征的关键,依然是分子“关闭”和“发光”间的切换。但在PALM显微镜下,分子会先后逐个发出荧光,从而实现特征区分。此外,分子不仅是一个一个地发光,而且发光顺序是随机的。但问题是,我们如何确定发光的分子位置呢?答案是,把相机当作探测器,当分子发出荧光、发射光子时,探测器就可以计算出光源的中心、即发射光子最多的地方,从而确定分子位置。所以该技术也是借助分子“开”和“关”的切换来区分特征的。

那么PALM/STORM显微镜与STED显微镜的区别究竟是什么?在STED显微镜中,我们需要让样本发出大量光子,才能决定哪些位置上的分子处于“开”状态,哪些处于“关”状态。而在PALM/STORM显微镜中,需要让探测器接收大量光子,才能计算出分子的中心位置。所以要想确定分子位置,都需要借助大量光子。
这一点非常重要。如果你是物理学家的话,就很容易明白为什么了。因为假如只有一个光子,它的运动就非常随机,你就无法确定光束的形状等等。但如果有大量光子,你就能更好地确定光束的位置。无论是对STED还是PALM/STORM,光子越多,定位就越精确。但这两类显微镜之间存在一大区别:在STED显微镜中,通过激光,已经有了大量光子;但在PALM显微镜中,光子则来自荧光染料,这样定位能力就比较受限了。
所以这也是STED的一大优势,因为由激光提供光子可以更精确地确定分子坐标。但另一方面,PALM/STORM显微镜可以实现更高的空间分辨率,这也是我的研究目标。因为PALM/STORM针对的是单个分子,这已经是高分辨率的最小单位了。如果能设法用较少的光子确定分子位置,想达到更多高分辨率就变得非常轻而易举了。
正如我上文所说,高分辨率的关键就在于分子“开”和“关”两种状态的切换。PALM/STORM显微镜的优势在于以单个分子为基础,虽然也存在耗时较长的缺点,但就空间分辨率来说,这是一大优势。但它的劣势在于,需要由染料提供大量光子。由于存在这一限制,所以空间分辨率仍难以降到几纳米的水平。相比之下,STED的优势在于对分子的定位很容易,因为激光可以提供大量光子。
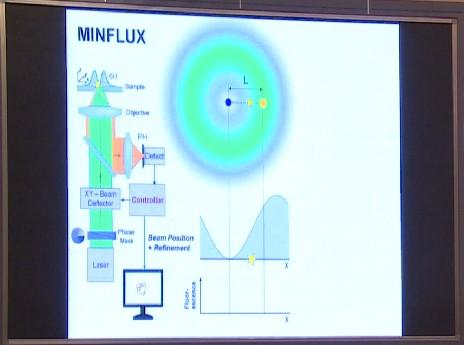
能否优势互补?
为何不把这两种显微镜的优势结合起来呢?一方面像STED一样,借助激光提供的光子实现轻松定位;另一方面像PALM/STORM显微镜一样,以单分子为基础。这样一来,就应当能实现高空间分辨率了。这便是MINFLUX的基本理念。
MINFLUX诞生
去年,我们发表了第一篇论文,后来又做了一系列相关研究。科学家对单分子、尤其是对荧光单分子的跟踪已经持续了很长时间。研究人员会为了各种各样的原因跟踪标记蛋白的运动。一般来说这会运用落射荧光照明技术,会照亮你的整个视野,然后把样本投射到相机上。相机会像旁边的监控屏幕上一样,产生一个衍射光斑。为确定分子位置,同样要计算出这个衍射光斑的中心坐标。这里同样是光子越多,定位就越精确。但它也同样受单个荧光分子发出的光子个数限制。如果光子过少,或者存在其它问题,就无法实现精准定位。
这里我们借鉴了STED的做法来实现精确定位,非常简单。我们不用落射荧光照明的模式,而是使用甜甜圈状的光束。这种形状的优势在于,它的中心点在样本空间中的坐标可以很容易地确定,并且这个“甜甜圈”越亮,中间的洞就越清楚。这在STED中也是同理。但和STED不同的是,这里“甜甜圈”处的光线为激发光线,可将分子切换到激发态。我们还能通过一种简单的方法,测试目标分子是否正好位于“甜甜圈”中心处:假如位于中心处,目标分子就不会发光;但如果目标分子偏离了中心点,就会和其它分子一样发光。
用这种方法,我们还可以有效追踪分子的位置。我们可以移动“甜甜圈”光束,只要目标分子不发光了,就能判断出该分子正好位于“甜甜圈”中心处;反之若目标分子继续发光,我们就要继续移动光束,直到该分子不发光为止。我喜欢用一个笑话来解释这个过程。假设有一个小魔鬼,它始终知道目标分子的位置,并且会据此移动光束,让目标分子始终与光束中心点吻合。这样一来,我们就能一直知道目标分子的位置。但由于目标分子始终位于光束中心点,它从始至终都不会发出荧光。所以这种方法无需发出一个光子,便能全程获知目标分子的位置。这与PALM/STORM和其它单分子追踪显微镜的定位方法有着根本性区别。这些方法都需要尽可能多的光子才能实现精确定位,而等这么多光子发射出来也需要很长时间。
而利用这种新方法,从理论上来说,由于小魔鬼知道目标分子在哪,我们不需要调整光束的位置,因此目标分子无需发射一个光子就能实现精确定位。但在现实中可没有这样的小魔鬼。我们需要用电子控制器来代替它的功能,控制光束偏转器、并且探测目标分子发出的光子。如果探测到了光子,就说明目标分子尚未处于“甜甜圈”光束的中心处。然后我们就可以据此做出调整。如果能使发出的荧光量最小化,就能精准确定目标分子的位置。换句话说,该技术不需要目标分子发出大量荧光光子,就能实现精确定位,因为定位所需的大多数光子其实已经由激光提供了,相当于用这道“甜甜圈”光束做了预定位,然后再根据目标分子的发光情况进行微调。这样一来,定位的压力就不像PALM/STORM显微镜那样、由目标分子发射的荧光承担,而是由“甜甜圈”光束来承担。这种方法效率很高,定位速度很快,对光子的利用率也很高,可以让高分辨率显微镜对分子进行快速追踪。
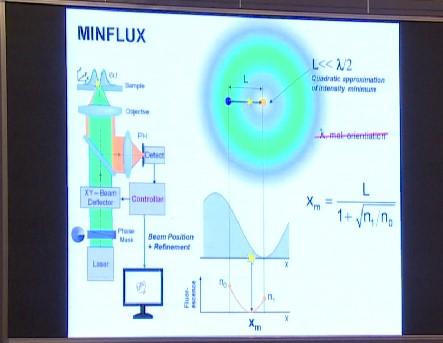
那么我们究竟是如何测定分子位置的呢?这里涉及很多技术上的细节。你们可以从中看出这套概念是多么重要。
现在我们来做一个思维实验。图上的黄色星星代表一个分子。假设我们想弄清这个分子在这条水平线上的位置。图片中间是“甜甜圈”光束的强度分布,最下面则是我们探测到的荧光强度。
随着光束移动,当目标分子刚好与光束中心重合时,就不会发射荧光。只要光束所处的位置没有发出荧光,就说明目标分子正好位于此处。就这么简单。你可能会认为,我们需要多次调整光束位置,才能确定分子位置。
但事实上,我们可以像PALM/STORM显微镜一样,用数学运算进行推断。假如光束在不同位置上的荧光强度能够连成一条抛物线,就能推断出零点所在的位置了。这就是个二次方程的问题,用中学代数知识就能解决。当然,你还要统计出接收到的光子数量,才能套用到泊松分布中去。这个等式包含的信息量很大。我们往往能从重要的现象中得出重要结论。这里的规律与波长无关。
这乍一听可能很令人吃惊,因为这毕竟是一台安装了透镜的聚焦光显微镜,竟然会与波长无关,无论是发射光还是激发光的波长都与之无关。这实际上意味着衍射障碍已经不存在了。由于定位不依赖分子朝向,这就解决了PALM/STORM显微镜存在的一大问题。PALM/STORM的分辨率之所以难以达到20纳米以上,就是因为存在朝向问题。而这些问题在这里都不复存在了,因为定位与发射朝向无关,而是完全由激光决定。
总结一下,这项新技术所需的光子更少,不取决于波长,且它的优势还不止于此。如果你对分子所在的位置有一个大致的概念,就可以把它放在“甜甜圈”光束下进行“放大”。这样一来需要的光子就更少了。而且这比收集更多光子的做法更高效。如果要拍三维图像,通常需要找四个点,但基本原理是相同的。其实一般测三个点就能确定某个分子的位置了。这种方法非常有效,也能节省所需的光子。假设你有10个光子,用MINFLUX就能达到10纳米的分辨率。但即便是用最完美的相机,也需要至少290个光子才能实现这一点。可见MINFLUX能够有效减少对光子的需求量,也能缩短记录时间。不仅如此,你还能把分子的估测位置进一步放大,从而更精确地确定位置。
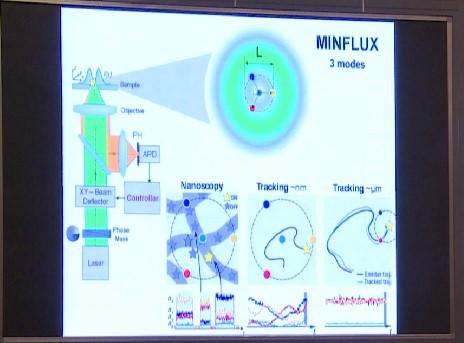
接下来,你可以像PALM/STORM显微镜一样,通过切换分子的开关状态进行成像处理;也可以进行有效的长距离或短距离分子跟踪。所以这是一种全新的分子定位方法。它可以用来观察活大肠杆菌体内核糖体的构成单元。这里的运动速度被放慢了50倍,相当于每秒进行8000次定位,分辨率约14纳米。我们还可以用该技术追踪分子的运动。假如分子固定不动,我们就可以精准确定它的位置。而如果分子会在一定范围内运动,我们便可以进行迅速跟踪。这种方法需要的光子数量只有传统方法的十七分之一,分辨率为2.5纳米,并且所需时间极短。

当然,这项技术还会不断改进。STED显微镜多年来一直在不断完善,MINFLUX也不例外,我相信它会带来很大的改变。这项技术本身就具有突破性。它从根本上改变了我们看待问题的方式。图片上这些分子之间仅相隔11纳米。若利用PALM/STORM显微镜,即使在最理想的情况下,也无法将它们分辨开来。但用MINFLUX就有可能,因为需要发射的光子更少,并且主要的定位工作是由“甜甜圈”激发光束完成的。我们不仅做了这一个实验。在另一项实验中,我们实现了6纳米分辨率,并且实验结果可复制。
整个研发过程可以说乐趣无穷。要提醒大家的是,我们虽然实现了8纳米、9纳米甚至6纳米的分辨率,但仍然是利用聚焦可见光和物镜完成的。若换在20年前,大家肯定会说,你不可能仅用普通的物镜和聚焦光线就把距离这么近的分子分辨开来。所以你如果问我接下来会发生什么事情,我会说,超分辨率显微镜的分辨率已经达到了真正极限、即分子直径,定位速度已经大大提高,但我们还未达到理想速度和光子数量下限,还需要进一步研发。你如果研究过分子定位问题,就会知道这些方法总有改进的空间。计算分子位置的中心点其实并不是超分辨率的关键要素。如果有人把这两者混为一谈,就说明他们并没有真正理解。

MINFLUX可以应用于多种领域。它可以替代荧光共振能量转移技术(简称FRET),实现高分辨率成像,比如按实际速度观察蛋白质的折叠过程等。当然,这些都要用到荧光标签。
从技术上来说,我们完全可以把整套装备放进一个鞋盒那么大的盒子里,并且能安装到任何显微镜镜架上。很多公司正在致力于实现这一点,未来,这将成为趋势。
《科学大家》专栏投稿邮箱:sciencetougao@sina.com 来稿请注明姓名、单位、职务




